Dans le cadre du programme 2 du SIRIC ILIAD, Jean-Baptiste Alberge, doctorant, s’est penché sur les modifications épigénétiques de l’ADN dans le myélome multiple, afin de mieux comprendre la maladie et son évolution.
Au sein de notre corps, toutes nos cellules possèdent les mêmes gènes. Ce qui détermine les différences entre une cellule nerveuse et une cellule de peau par exemple, réside notamment dans le niveau d’expression de ces gènes. L’expression plus ou moins importante, est déterminée par différents facteurs et notamment par des modifications épigénétiques. Ces modifications peuvent par exemple être portées par la chromatine ou la molécule d’ADN en elle-même.
C’est cette thématique que Jean-Baptiste Alberge, doctorant au sein du programme Pister, a étudié dans le cadre de sa thèse, afin de mieux comprendre le myélome multiple, cancer rare de la moelle osseuse qui touche 5 personnes sur 100 000 en Europe. Il s’est plus particulièrement intéressé à une marque chimique de l’ADN la 5-hydroxyméthylcytosine (5hmc), une hydroxyméthylation de l’ADN.
L’étude de la 5hmc devait relever un défi technologique : permettre à l’équipe nantaise d’étudier les zones actives de la chromatine, pour des échantillons d’ADN extraits sans chromatine. L’idée était de montrer que la présence de la 5hmc était suffisante pour retrouver les zones actives de la chromatine déjà connues et d’en découvrir de nouvelles. L’objectif était de cartographier l’activité de la chromatine sur le génome et cela en l’absence de chromatine. Les chercheurs ont ainsi quantifié la 5hmc pour le myélome multiple et ont pu étudier les régions actives de la chromatine.
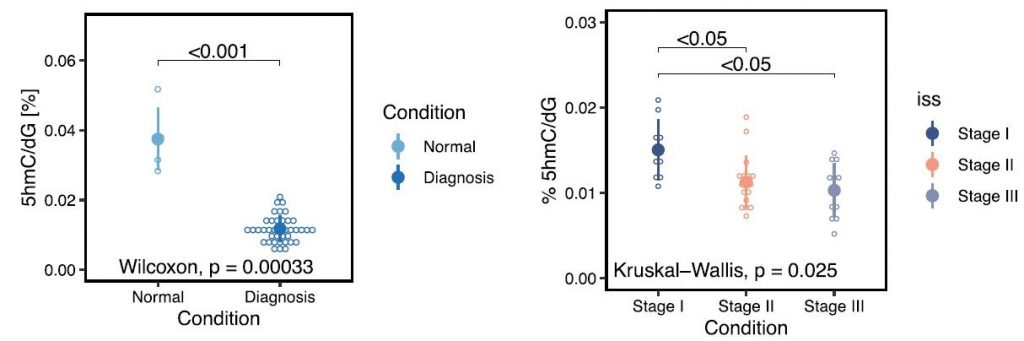
Si ces échantillons d’ADN ont rendu plus compliquée l’étude de la chromatine, ils ont néanmoins apporté du recul sur la maladie. Collectés en 2009, soit 10 ans avant ces travaux, ils ont permis aux chercheurs d’associer aujourd’hui la présence de la 5hmc à des informations sur la rechute et l’évolution de la maladie. L’équipe de recherche a ainsi montré que les personnes atteintes du myélome multiple ont 3 fois moins de 5hmc que les personnes non malades. Et plus encore, plus le myélome multiple est à un stade avancé, moins il y a de 5hmc dans le génome (voir image ci-dessous). Le niveau de 5hmC pourrait donc permettre d’évaluer la sévérité de la maladie grâce à l’analyse de l’ADN génomique. Ces résultats sont néanmoins préliminaires et demandent des études supplémentaires pour être confirmés et complétés.
Lire la publication scientifique
Modifications épigénétiques : Modifications qui régulent l’expression des gènes, transmissibles par division cellulaire, mais qui ne sont pas portés par la séquence génétique elle-même (= séquence de nucléotides).
Chromatine : La chromatine est constituée d’ADN compacté mais aussi d’ARN et de protéines qui structurent et permettent à l’ADN de se compacter. Parmi ces protéines on retrouve les histones, autour desquelles la molécule d’ADN s’enroule.
Hydroxyméthylation : Ajout d’un groupement OH (hydroxy) + CH3 (méthyl)
5-hydroxyméthylcytosine (5hmc) : Ajout d’un groupement OH (hydroxy) + CH3 (méthyl) sur une cytosine (C)









